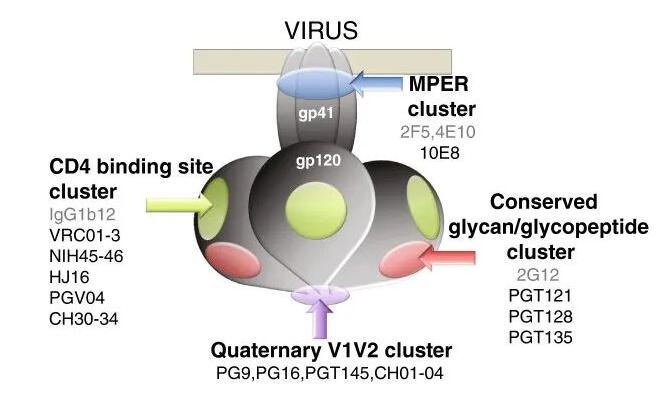
研究发现:HIV表面存超快运动元素,为疫苗研发开辟新路径
发布日期:2024-03-12
点击:次
 |
文章原名为“微秒动力学控制HIV-1包膜构象”,作者为Ashley L. Bennett等人。文章于2024年2月2日发布在《Science advances》上,《Science Advances》作为《Science》杂志的姊妹期刊,它是由美国科学促进会(AAAS)出版的一份同行评审的开放获取科学期刊,自2015年开始发行,旨在提供一个快速发布创新和重要科学发现的平台。该期刊支持跨学科研究,鼓励提交具有突破性、广泛影响的研究成果。
本文介绍了一系列高级生物技术实验,包括冷冻电子显微镜(冷冻电镜)、静态和时间分辨小角X射线散射(SAXS)技术,以及分子动力学模拟,旨在深入研究HIV病毒的信封蛋白(Env)SOSIP三聚体的结构和动态。通过对CH505和CH848 Env SOSIP变体在不同构象下的详细分析,研究团队揭示了这些复杂蛋白质分子如何响应温度变化和糖基化的影响,以及它们如何与抗体结合。
此外,文中还描述了基于结构的设计方法来优化SOSIP三聚体的表达和稳定性,增强其与特定抗体的结合能力。这些研究对于理解HIV病毒的感染机制、设计更有效的疫苗以及开发针对HIV的治疗策略具有重要意义。
HIV-1 的 Env 糖蛋白可以引发一系列复杂的结构变化,帮助病毒与宿主细胞融合。虽然科学家们已经对 Env 糖蛋白可能采取的不同形态有了较深入的理解,但关于这些变化发生的微秒级速度的研究还很少。
我们采用了一种特殊的 X 射线散射技术,能够以微秒级的精确度观察 HIV-1 Env 结构如何在极短的时间内重新排列自身。在研究中,我们发现了两种 Env 变种在数百微秒内完成的结构转变,这与之前已知的 Env 结构变化相吻合。
更让人惊讶的是,我们还发现了一个全新的结构变化,发生时间不到 10 微秒,这一变化涉及到病毒表面三聚体顶部从有序结构向无序结构的转换。基于这些发现,我们设计了一种新的 Env 结构,通过在相邻的病毒蛋白之间形成二硫键,将病毒固定在接近融合前的闭合状态。
这项研究揭示了微秒级别的结构动态对于病毒如何控制其表面蛋白形态至关重要,这对未来的疫苗开发具有重要意义。
HIV-1 的 Env 糖蛋白是病毒攻击宿主细胞的“前锋”,它能够帮助病毒附着并进入宿主细胞内部。这个过程涉及到 Env 糖蛋白结构上一连串复杂的变化,这些变化是由特定的受体 CD4 和共受体 CCR5/CXCR4 触发的,最终实现病毒与宿主细胞的融合,导致宿主细胞被感染。
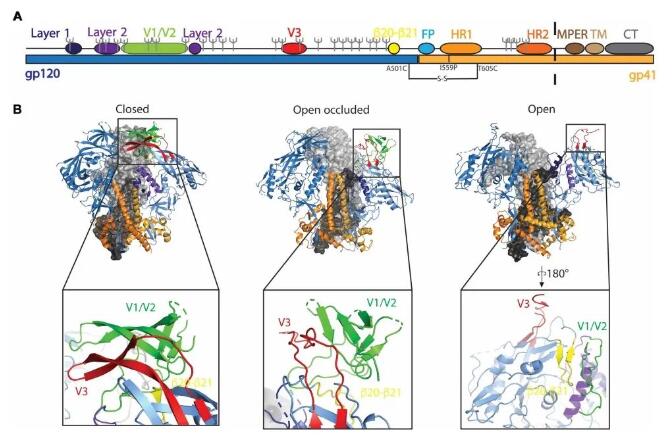
Env 糖蛋白由三部分不同的原型体组成,每个部分包括一个可以与受体结合的 gp120 部分和一个负责融合过程的 gp41 部分。在病毒准备融合前,这些 gp120 部分会紧紧包裹住 gp41 部分,防止过早暴露融合机制。当 CD4 受体结合到 Env 糖蛋白上时,会引发 gp120 部分的内部重排和旋转,使得 gp41 的融合部位暴露出来。
通过高级显微技术,科学家们已经明确了 Env 糖蛋白在这一过程中的关键结构特征,这些特征控制着 Env 糖蛋白的动作。当病毒处于准备融合的闭合状态时,不同 gp120 部分之间以及它们与 gp41 部分之间的联系会保持稳定。
但是,一旦 CD4 受体结合,这些联系就会被打断,Env 糖蛋白转变为开放状态,准备与宿主细胞融合。虽然目前我们对 Env 糖蛋白从闭合到开放状态转变的终点结构有了较清晰的认识,但其间的具体转变过程仍然不甚明了。
当 HIV-1 病毒的 Env 糖蛋白从准备融合的闭合状态变为开放状态时,会发生一个关键的变化:两个变异环节V1/V2和V3之间的顶点连接会被切断。在这个过程的最终阶段,当 Env 糖蛋白同时绑定到 CD4 受体和一个模仿共受体的抗体上时,我们可以看到 gp120 部分发生旋转,V3 环变得暴露和灵活,而 V1/V2 部分则发生了动态的构象变化。
有趣的是,当 Env 糖蛋白与一个广泛中和的抗体 b12 相互作用时,虽然也观察到 gp120 的旋转,但 V1/V2 和 V3 并没有发生重大的结构变化。
科学家们还发现了一些其他的结构,这些结构中 gp120 发生了旋转,但 V1/V2 被遮挡,形成了一种被称为“开放-遮挡”的状态,这可能是 Env 糖蛋白从闭合状态向开放状态转变过程中的一个中间阶段。
通过使用单分子 Förster 共振能量转移(smFRET)、双电子-电子共振(DEER)和氢-氘交换质谱等高精度实验技术,研究人员发现,即使在没有受体结合的情况下,Env 糖蛋白的结构也是非常动态的。然而,将这些实验观察与已知的 Env 结构联系起来还面临着诸多挑战,特别是这些技术目前还难以探测到控制 Env 结构变化的微秒级时间尺度的细节。
在开发 HIV-1 疫苗时,深入了解 Env 糖蛋白的结构和动态变化是至关重要的。研究人员认为,阻止 Env 糖蛋白从准备融合的闭合状态转变为开放状态对于筛选出能够通过疫苗接种产生有效中和反应的广泛中和抗体至关重要。Env 糖蛋白是疫苗设计努力的焦点,这包括研究与细胞膜相关的完整 gp160 蛋白和一种被截短的可溶性 gp140 结构。
为了增强这些 Env 结构的稳定性,科学家们常常使用特定的突变,比如在 gp120 和 gp41 之间引入二硫键(SOS突变)和将 Ile559 替换为 Pro(IP突变),以此分别防止 gp120 的脱落和阻止它从准备融合状态发生变化。
此外,为了确保 gp41 的融合元素不会被暴露,对这些所谓的 SOSIP 免疫原进行额外的稳定化处理被证明是必需的。这包括对 gp120 和 gp41 之间,以及三聚体基部的原型体内和原型体间接触进行基于结构的设计改进。
尽管采取了这些额外的稳定措施,疫苗接种有时仍可能诱发效果不佳的针对特定表位的抗体。虽然已有的单分子 Förster 共振能量转移(smFRET)和氢-氘交换质谱(HDX)实验能够描述 Env 在从毫秒到小时不等的时间尺度上的动态变化,但我们对于控制这些变化的微秒级时间尺度的结构动态知之甚少。因此,更深入地理解 Env 从闭合到开放状态的转变路径,将为设计更稳定的闭合状态 Env 三聚体提供宝贵的指导。
在这项研究中,我们利用了两种变种的 HIV-1 Env 结构,CH505 和 CH848,通过特殊的小角X射线散射实验探索 Env 蛋白如何在微秒级时间尺度上从闭合状态转变为开放状态。这些 Env 结构被设计成包含一个在特定位置(201C和433C)形成的稳定化二硫键,以增强其稳定性。
我们发现,CH505变体中一个特定的结构中间体能够在大约7微秒内迅速形成,并在大约466微秒内转变到另一个明显不同的结构状态。相比之下,CH848变体的转变速度更快,显示出大约2微秒和200微秒的时间常数,这表明尽管CH848变体在热稳定性和抵抗开放状态靶向抗体结合方面表现得更好,但其转变速度却更快。
通过对这些实验数据进行结构建模,我们认为这些结构变化涉及到 Env 三聚体顶部的 gp120 蛋白之间的连接断开,紧接着是 gp120 蛋白向外旋转,远离中心轴。基于这一发现,我们设计了一种新型的 Env 结构,通过在顶点处“钉合”原型体间的连接,阻止了对第一个迅速形成的中间体的访问。
使用这种稳定化结构与特定抗体结合的实验结果显示,我们能够保持 Env 蛋白在接近融合前的闭合状态,其中一个抗体b12在结合角度上与未稳定化的 Env 结构相比,显示出了显著的变化。
这项研究的结果强调了了解 Env 结构在转变过程中的瞬态中间状态对于指导疫苗设计的重要性,为开发能够更有效阻止 HIV-1 病毒入侵宿主细胞的疫苗提供了新的思路。
图1 HIV-1 的 Env 糖蛋白是一个结构多变的分子。(A) 在 HIV-1 Env 的结构图中,gp120 部分用蓝色标出,而 gp41 部分则用浅橙色标出。这个结构图还特别标示了多个关键的结构元素,包括两个不同层次的区域(深蓝和紫色)、两个变异域(V1/V2用绿色表示,V3用红色表示)、以及其他几个特定的区域(比如融合肽以青色表示,七联重复序列以橙色和深橙色表示等)。此外,图中还用灰色的小叉表示了 CH505 病毒的糖基化位点,而一个黑色的虚线则显示了 SOSIP 构造在哪里被截断。(B) 从侧面看,图中展示了三种不同状态的 Env 三聚体的结构:前融合闭合状态(左边,PDB ID 6UDA)、开放-遮挡状态(中间,PDB ID 5VN8)和完全开放状态(右边,PDB ID 5VN3)。每个结构中的 gp120 和 gp41 部分按照(A)部分的颜色编码进行标记。另外,还有小图展示了每个状态下 V1/V2、V3 和 β20-β21 元素的详细放大视图,以便更清楚地看到这些关键区域的变化。
SAXS 捕获 HIV-1 Env 糖蛋白的构象转变
在我们的研究中,我们利用了一种叫做 SAXS 的技术来观察 HIV-1 Env 糖蛋白在结构上的变化。这种实验方法包括两个步骤:首先是“泵送”阶段,我们通过一个红外激光快速加热样品周围的水,这样做是为了打乱系统的平衡状态;随后是“探测”阶段,在这个阶段我们测量系统在向新平衡状态过渡过程中的状态。为了成功地实现这种方法,我们需要一个能够在温度突变(T-Jump)下轻松改变构形的 Env 构造,并且这种构形的变化可以通过 SAXS 技术检测到。
因此,我们首先寻找一个适合进行这种 SAXS 实验的 Env 构造。我们利用一种叫做生物层干涉测量(BLI)的技术,来测试两种特殊稳定化的 Env 结构(分别来自 BG505 和 CH505 病毒)与特定抗体的结合情况。这些抗体分别针对 Env 的闭合状态和开放状态。
我们发现,尽管某些 HIV-1 病毒的 Env 蛋白在没有 CD4 受体的情况下就能变为开放状态并结合到特定抗体上,但是在我们的实验中,只有 CH505 的 Env 结构表现出了与这些抗体的结合,而 BG505 的则没有。这表明 CH505 的 Env 结构更加易于在实验中的温度变化下发生构形变化。
通过这些实验,我们确认了两种 Env 结构在闭合状态下都能够结合到一种特定的抗体(PGT145),这验证了它们闭合状态的存在。同时,CH505 的 Env 结构还能与另外两种针对开放状态的抗体进行交互,表明它在结构上更加灵活,可能更适合于我们探测快速时间尺度上结构重排的实验目的。
在用 SAXS 技术研究溶液中的蛋白质时,由于蛋白分子运动造成的散射是均匀的,这让我们能够得到关于蛋白质大小和总体形状的基本信息。通过识别与蛋白质特定结构变化相关的散射曲线上的特定特征,我们可以更好地理解蛋白质的结构变化。
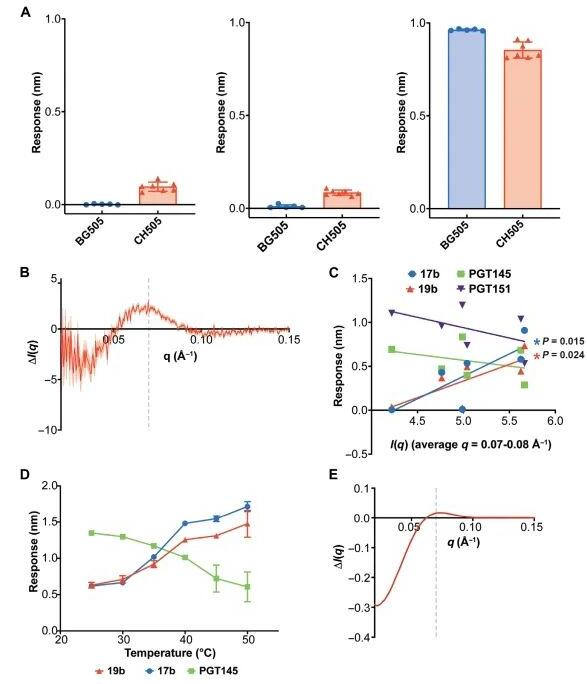
考虑到 CH505 和 BG505 这两种 Env 结构在与特定形态抗体的结合测试中表现出的差异(如图 2A 所示),我们预期它们的 SAXS 散射图谱中会有明显的不同。因此,我们分别测量了 CH505 和 BG505 的 SAXS 散射图,并通过相互比较,计算出了它们之间的差异(如图 2B 所示)。
这种差异图显示出一个特定的散射峰值,在散射向量(q)值0.07 Å−1处,这个峰值先是在0.01到0.05 Å−1区间下降,然后在0.00到0.01 Å−1区间上升。
接下来,我们探索了这一 SAXS 特征是否与 CH505 衍生的 Env 结构在特定构型下与抗体的结合有关。这包括了多个不同时间点分离出的 CH505 变体、一个特别稳定化处理的 CH505 以及 BG505。
我们为每一种 Env 结构测量了 SAXS 散射图,并通过生物层干涉技术(BLI)测试了它们与抗体17b、19b、PGT145和PGT151的结合情况。这项测试帮助我们更深入地理解了这些 Env 结构如何响应结构上的变化,并在结构变化与抗体结合之间建立了联系。
通过对一系列数据进行线性回归分析,我们发现 SAXS 技术测得的数据与两种抗体(17b 和 19b)的结合反应之间存在显著的相关性,这一发现的统计显著性分别为 P = 0.015 和 0.024(如图 2C 所示)。
这说明 SAXS 技术能够有效捕捉到与这些抗体结合相关的结构变化。然而,对于另外两种抗体(PGT145 和 PGT151),尽管我们也观察到了一种趋势,即它们的结合反应与 SAXS 数据呈现负相关,这种趋势符合闭合状态与开放状态 Env 结构的区别,但这种相关性并不显著(P = 0.127 和 0.270)。
这种缺乏显著相关性可能表明还有其他因素在影响抗体的结合,这些因素在 SAXS 的测量结果中没有被直接反映出来。特别是,PGT145 和 PGT151 需要穿过 Env 结构上的糖基护盾才能结合到相应的表位,而 17b 和 19b 则不需要这样做。
这意味着不同 Env 变体之间的糖基护盾差异可能是影响这两种抗体结合相关性的一个因素。总的来说,这些结果展示了 SAXS 技术在报告 SOSIP 糖蛋白间已知结构状态差异方面的有效性。
在我们的研究中,我们利用了一种名为奇异值分解(SVD)的方法,来处理静态 SAXS 数据中的曲线,目的是更清晰地分析数据。SVD 是一种数学技巧,它能够将复杂的数据集拆分成几个简单的矩阵:组分曲线(或我们可以称之为“左向量”)、奇异值,以及它们的贡献(或“右向量”)。
通过将这些矩阵相乘,我们能够重建原始的数据曲线。这个过程不仅帮助我们减少了数据中的噪声,还能将信号分解成不同的部分,这些部分可能与我们感兴趣的特定物理特性有关,例如 Env 结构倾向于处于开放状态的能力。
在这项分析中,我们的输入数据是 SAXS 强度的一个矩阵,输出则是一系列帮助我们分离信号的向量。通过这种方式,我们有效地从数据中去除了噪声,并分离出了代表不同过程的信号。
值得注意的是,SVD 并不总是将信号分解为有直接物理意义的向量。例如,我们发现的第一个左向量在 0.02 到 0.05 Å−1 区间内有一个负峰,以及在 0.07 Å−1 有一个较小的正峰。第二个左向量则显示了与 BG505 和 CH505 之间的差异曲线相反的特征,暗示这可能与开放状态或类似开放状态的倾向有关。
总的来说,这些发现揭示了不同的 Env 结构在 SAXS 曲线上呈现出的差异,特别是在 0.07 Å−1 的峰值处,这些差异可以告诉我们 SOSIP 结构倾向于处于何种状态——无论是开放状态还是某种类似开放的状态。
HIV-1 Env 糖蛋白的构象转变可以通过提高温度来诱导
通过提升温度,科学家们发现可以促使 HIV-1 的 Env 糖蛋白发生形态上的变化。在这个过程中,他们利用了一种名为 TR, T-Jump SAXS 的特殊实验方法,该方法通过快速的红外激光脉冲来引起温度的突然跳变,进而影响蛋白质的结构平衡。
实验中,研究人员特别选用了 CH505 SOSIP 这种 Env 结构,因为它不仅结构上比较容易发生变化,而且即便在没有 CD4 激活的情况下也能自然地转变到所谓的“开放状态”。为了探究温度变化对这种 Env 结构动态的影响,他们在从 25°C 到 50°C 不等的多个温度点上测量了 CH505 SOSIP 与三种抗体(17b、19b 和 PGT145)的结合情况。
结果显示,随着温度的上升,17b 和 19b 与 Env 结构的结合增多,而 PGT145 的结合则减少,这说明在较高温度下,趋向于开放状态或类似开放状态的 Env 结构比例有所上升。
然后,我们探索了 SAXS 技术是否能够检测到因温度升高而引起的 CH505 SOSIP Env 结构的变化。通过在不同温度点(25°、35°、40°、44° 和 50°C)对 CH505 SOSIP 进行静态 SAXS 散射轮廓的测量(如图 S3 所示),我们发现了一个有趣的现象:在 0.07 Å−1 的差异轮廓中,出现了与 CH505 和 BG505 SOSIP 之间差异相同的特征(如图 2E 所示)。
与 CH505 和 BG505 在 25°C 时的差异轮廓不同(如图 2B 所示),我们观察到在散射向量值小于 0.03 Å−1 时曲线呈现下降趋势(如图 2E 所示)。
为了更深入地理解这些变化,我们对静态温度序列的数据集进行了奇异值分解(SVD),分析了那些定义信号的主要组分。这些分析结果的第一和第二组分与之前的静态 SAXS SOSIP 数据集中的结果相似(如图 S3H 所示),表明 CH505 的结构对温度非常敏感,随着温度的升高,其结构的“开放性”也随之增加。总的来说,这些发现证实了 CH505 SOSIP 的结构变化对温度十分敏感,且这种变化是可以通过 SAXS 技术进行有效测量的。
图2 静态 SAXS 散射轮廓揭示了 HIV-1 Env 蛋白的形态变化。(A)对于 BG505 SOSIP(用蓝色圈表示)和 CH505 SOSIP(用红色三角形表示),通过生物层干涉测量(BLI)得到的与抗体 17b、19b 和 PGT145 结合的反应。误差条展示了基于五个和七个分别独立制备的 BG505 和 CH505 SOSIP 批次计算得到的平均值的标准偏差。(B)展示 BG505 和 CH505 散射数据之间差异的静态 SAXS 散射差异曲线。曲线中的阴影区域表示误差的范围。在散射差异特征(q)的峰值约 0.07 Å−1 处有一条灰色虚线标出。(C)在 0.07 至 0.08 Å−1 范围内 SAXS 散射强度平均值与一系列 SOSIP Env 蛋白与 17b、19b、PGT145 和 PGT151 抗体结合的反应之间的关系,分别用蓝色圈、红色三角形、绿色方块和紫色倒三角形表示。(D)在不同温度下 CH505 Env SOSIP 与 17b、19b 和 PGT145 抗体的结合反应,误差条表示三次实验的平均值的标准偏差。(E)通过将 CH505 Env SOSIP 在 25°C 时的 SAXS 散射数据与 50°C 时的数据相减,得到的散射差异曲线。这个差异在 q = 0.07 Å−1 处有一个明显的峰值,由灰色虚线标示。
TR, T-Jump SAXS 揭示了 CH505 SOSIP SAXS 轮廓的时间依赖变化
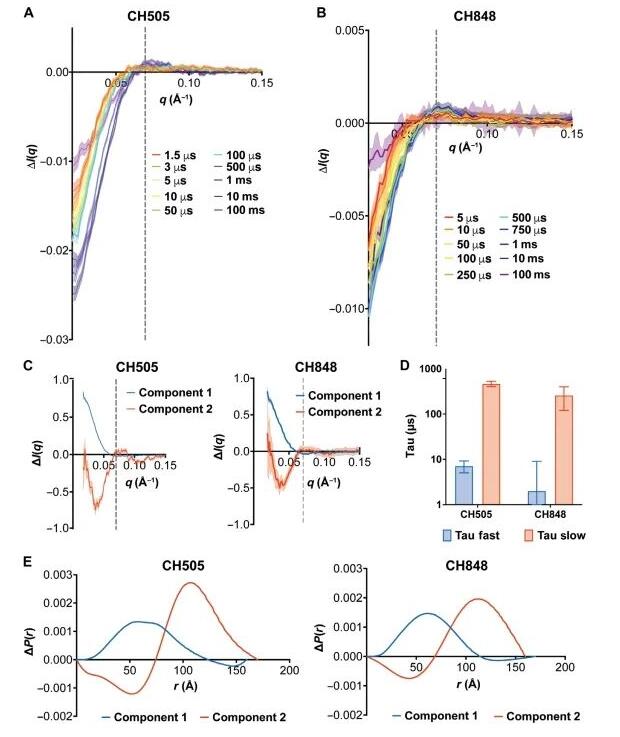
通过 TR, T-Jump SAXS 技术,我们进一步探究了 CH505 SOSIP 在快速加热后的时间相关结构变化。首先,我们确定了实验中使用的最佳初始平衡温度。根据之前的实验,我们选择了从 40°、42°、44° 和 46°C 开始的温度跳变,并设置了 10 微秒和 1 毫秒的探测延迟。结果显示,在 44°C 时,我们关注的区域(0.02 到 0.10 Å−1)内的信号差异达到最大。进一步分析确定,当温度从 44°C 跳变时,系统的加热程度大约为 6°C,使系统的最终温度约为 50°C。
随后,为了详细了解加热后 CH505 SOSIP 结构随时间的变化,我们在多个不同的延迟时间点(从 500 纳秒到 100 毫秒)测量了散射数据。最初,在 500 纳秒的测量中,我们观察到 0.02 到 0.05 Å−1 区间内一个显著的负峰,表明有一个非常快速的变化过程。随着时间延长,这个特征变得更加明显,尤其是在 1.5 到 3 微秒之间的增强显著。此外,随着延时增加,在 q = 0.07 Å−1 的位置出现了第二个更为明显的信号。这些发现与我们对静态 SAXS 数据的分析相吻合,显示了类似的结构变化。
在更长的时间延迟(10 毫秒和 100 毫秒)观察到,随着系统温度逐渐回归初始状态,这些结构差异开始减小,表明 Env 结构逐步恢复到初始的平衡状态。这些结果展示了 CH505 SOSIP 结构对温度的敏感性以及随时间变化的动态过程,能够通过 SAXS 技术精准捕捉。
这些研究结果共同证明了 CH505 Env 结构在微秒级时间范围内可以发生可逆的结构变化。随后,我们探究了一个更稳定的 Env 变体是否也会表现出类似的结构转变。我们对一个设计了二硫键以阻止其向开放状态转变的 CH848 Env 变体 SOSIP 三聚体进行了 TR, T-Jump SAXS 测试。
与 CH505 SOSIP 三聚体不同的是,CH848 变体并没有表现出对开放状态特定抗体 17b 的结合能力,并且对 19b 的结合也明显减弱,这暗示 CH848 三聚体进入开放状态的能力较弱(参见图 S1C)。热变性实验进一步证实了 CH848 SOSIP 三聚体比 CH505 SOSIP 三聚体更为稳定,变性转变的温度分别为 73.6°C 和 68.9°C。
通过在不同的时间延迟(从 5 微秒到 100 毫秒)收集的 TR, T-Jump SAXS 数据,我们观察到了类似于在 CH505 实验中所看到的 SAXS 差异(参见图 3B 和图 S6)。因此,尽管 CH848 三聚体相比 CH505 三聚体显示出更高的稳定性和较低的开放状态抗体结合能力,这些结果表明 CH848 在所测量的时间范围内同样经历了相似的结构变化。这些发现强调了即使是在不同稳定性水平的 Env 变体中,结构转变的基本机制仍然相似。
图3通过高级技术研究HIV-1病毒外壳蛋白,科学家们发现了两个关键的结构变化过程。(A) 对CH505病毒样本进行的测试显示,在不同的短时间内(从1.5微秒到100毫秒),病毒外壳蛋白的结构有显著变化,这些变化通过一系列颜色(从红色到洋红色)标示出来。(B) 另一种病毒样本CH848的测试也显示了相似的变化,时间延迟从5微秒到100毫秒不等。这些变化点位于图表的特定区域,通过灰色虚线标出。图表中的阴影部分展示了测量数据的标准误差,即数据的准确性水平。(C) 进一步的分析揭示了两种主要的变化组件,一种是快速变化(蓝色),另一种是慢速变化(红色),这两种变化在两种不同的病毒样本中都被观察到。(D) 对这些变化进行数学建模分析,确认了病毒结构变化的快速和慢速阶段。(E) 最后,通过对比两种病毒样本的结构差异,研究人员能够更深入地了解病毒如何在不同条件下调整其结构,这对于未来开发针对HIV的疫苗和治疗方法具有重要意义。
Env 通过快速形成的中间体转变
然后,我们进一步分析了 CH505 和 CH848 两种 Env 三聚体在 TR, T-Jump SAXS 实验中经历的结构转变。通过奇异值分解(SVD)技术,我们提取了这些数据集中的关键组分曲线。我们特别关注了 1.5 微秒至 1 毫秒这一时间段内的差异曲线,这段时间内的数据能够反映出在样品开始冷却之前发生的结构变化。
分析结果表明,之前在 CH505 和 BG505 SOSIP 的静态 SAXS 数据中发现的两个主要组分,在 CH505(参见图 S8B)和 CH848(参见图 S9B)的数据中也有所体现。对于 CH505 数据,我们注意到第一个主要组分在较短和较长的延迟时间内都发生了变化,而第二个组分的变化则主要出现在较长的延迟时间(参见图 S8D)。
此外,从第三到第八个 SVD 组件的数据显示,这些组分的变化在零点附近随机波动,说明它们对整体的 TR SAXS 信号贡献较小。而对 CH848 数据的 SVD 分析则表明,虽然存在相同的两个主要组分,但我们只能通过直观观察确定在这两个组分中各自发生了一次明显的变化。
随后,我们利用动力学模型来分析 CH505 和 CH848 两种 Env 三聚体的数据,特别是针对它们的前两个主要变化组分。通过对 CH505 的第一个主要变化组分应用双指数模型拟合,我们得到了两个不同的时间常数:7 ± 2 微秒和 466 ± 63 微秒,这些数据显示在图 3D 和表 S4 中。对于第二个主要变化组分,我们使用单指数模型拟合,得到了一个时间常数为 789 ± 183 微秒(同样展示在图 3D 和表 S4 中)。
接着,我们对 CH505 数据在 0.02 到 0.1 Å−1 区间内的曲线下面积(AUC)应用了双指数模型,得到的结果与前面两个时间常数 7 ± 2 微秒和 502 ± 40 微秒相吻合,这验证了我们的两次转变模型的准确性(见图 S8H 和表 S4)。
对于 CH848 的分析,我们发现第一和第二个主要变化组分的单指数拟合显示时间常数分别为 213 ± 69 微秒和 110 ± 15 微秒,这表明 CH848 的结构重排也在类似的时间尺度内发生(如图 3D、图 S9D 和表 S4 所示)。而基于 CH848 曲线下面积的计算揭示了一个单一转变,时间常数为 197 ± 55 微秒,与之前 SVD 分析得到的时间尺度保持一致(参见图 S9H 和表 S4)。
这些分析结果进一步证实了 CH505 和 CH848 Env 三聚体结构变化的动态特性,以及我们用于捕捉这些变化的方法的有效性。
在研究中,我们发现 CH505 Env 三聚体的一个关键部分在两个不同的速度上进行着变化。更慢的变化与另一个部分的变化同时进行,说明这些结构变化是同步发生的。值得一提的是,这些变化并不意味着每次都是相同的结构改变。
对 CH848 的分析显示,其结构变化的速度与 CH505 的第二部分非常相似,这一点从它们的动力学模型中可以看出。特别地,我们发现 CH848 的一个部分在较慢的速度上发生变化,与 CH505 中的某些慢速变化一致。这暗示了我们使用的双指数模型能够有效地捕捉到这一缓慢的过程。然而,由于缺乏在特定时间范围(1到5微秒)内的数据,这个模型的拟合并不完美。
不过,这个模型支持了一个观点,即 CH848 中的某些变化可能比 CH505 中相似的快速变化发生得更快。为了更准确地验证这一模型,我们需要在 1 到 5 微秒的时间范围内收集更多数据。尽管如此,我们在 CH505 中观察到的快速形成的中间态很可能也存在于 CH848 中。
总的来说,这些发现表明,这两种 Env 三聚体都能够迅速地过渡到开放或类似开放的状态,而且这两种 SOSIP 以不同的时间常数进行转变。
奇异值分解(SVD)技术不一定能将数据拆分为有实际物理意义的部分。为了解决这一点,科学家们开发了一种新的算法,名为 REGALS,这个算法通过对 SVD 分解过程加入实验上确定的限制条件,使得从 SAXS 数据集中解析出的各个组分在物理上更加贴近现实。我们应用了 REGALS 解卷积方法来分析 CH505 和 CH848 SOSIP 的时间分辨 SAXS 差异数据,目的是精确识别出这些数据中的独立散射组分(见图 3C,以及附加图 S8、F和G,S9、F和G)。
在这个过程中,我们参照了之前通过 SVD 和曲线下面积(AUC)分析确定的两个主要组分,为 CH505 和 CH848 分别设定了最小尺寸为 154 和 160 埃,以及最大尺寸为 173 和 162 埃。这些尺寸的设定是基于对 Env 结构模型的尺寸分析以及对模型的迭代优化过程得出的。
通过对数据进行拟合,我们为 CH505 和 CH848 分别得到了大约 1.0 和 0.124 的整体 χ2 值,这表明了我们的模型拟合效果良好。值得注意的是,每一个通过 REGALS 算法得到的组分都与其对应的 SVD 组件在特性上相似,尽管它们在图中关于 x 轴呈现镜像关系。
REGALS 分析不仅帮助我们将散射差异数据细分成独立的组分,还提供了一种用于描述这些组分中粒子原子间距离变化的配对距离分布差异(见图 3E)。在 CH505 和 CH848 的分析中,特别是对第二组分的距离分布差异分析揭示了结构变化覆盖了一个较大的范围,最大可达约 150 埃。
具体来说,CH505 和 CH848 的第二组分在 0 到 75 埃区间内的成对距离显著减少,而在 75 到 150 埃区间内的距离则相应增加。对于第一组分,最显著的距离变化发生在更小的范围内,平均距离约为 60 埃。
进一步的结果分析表明,在时间延迟增加时,CH505 的第二组分浓度上升,而第一组分在早期和较长时间的延迟都有所下降。相比之下,CH848 的数据显示,在较长的时间延迟中,第一组分的浓度增加,而第二组分则减少。这些观察结果与我们通过 SVD 右向量分析得出的组分贡献变化一致。
总体而言,REGALS 和 SVD 的联合分析为我们提供了一个包含两种不同结构变化的模型,这些变化分别发生在 CH505 和 CH848 中。这些距离分布的信息为基于结构的建模提供了宝贵的数据支持,帮助我们更深入地理解了这些 Env 三聚体在结构上的动态变化。
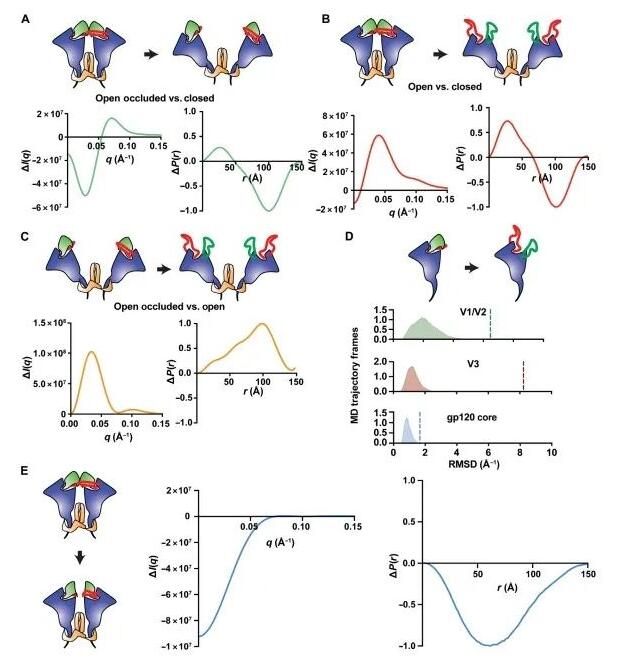
图4 快速和慢速过程分别代表两种独特的结构变化。(A 至 E)通过简化图解展示了 Env 蛋白不同的结构状态,涉及的关键区域有 V1/V2(绿色)、V3(红色)、gp120(蓝色)和 gp41(橙色)。(A)从闭合状态过渡到半开放状态时,理论上 SAXS 的差异曲线(图左下方)及其配对距离分布的差异(图右下方)。(B)从闭合状态转变到完全开放构形的理论散射差异曲线(图左下方)和配对距离分布的差异(图右下方)。(C)从半开放状态到完全开放状态的转变,其理论 SAXS 散射差异曲线(图左下方)和配对距离分布的差异(图左下方)。(D)在 gp120 单体中 V1/V2 和 V3 区域重排的示意图(图顶部)。对于250个5微秒的MD模拟,每个模拟包含1000帧,其中 V1/V2(绿色)、V3(红色)和 gp120 核心部分(蓝色)β-片层 α-碳的根均方偏差(RMSD)显示在图底部。虚线表示在闭合与开放构形间这些区域的 RMSD 差异。(E)顶部结构从有序到无序转变时的理论 SAXS 散射差异曲线(图中间)和配对距离分布的差异曲线(图右侧)。这些分析结果帮助我们理解 Env 蛋白在不同状态下的结构特征及其转变过程。
较慢的转变涉及开放闭塞状态的形成
我们进一步研究了 CH505 和 CH848 SOSIP 所展现的结构转变及其状态与已知的 HIV-1 Env 超域结构和动态之间的联系。先前的单分子荧光共振能量转移(smFRET)研究揭示,一个非对称中间态能够连接 Env 超域的闭合状态和开放状态(17–20)。
这些研究表明,从基本状态到非对称中间态,以及从非对称中间态到开放状态的转变可能在测量的毫秒级“死区”内发生,这与我们当前研究中的发现相一致(见图 3D 和表 S4)。非对称中间态的特征是其中一个原型体呈开放状态,而另外两个原型体则保持一种不同的构形。
结合我们观察到的,随着温度升高,针对开放状态的抗体结合增加以及静态 SAXS 散射数据的变化(见图 1C),我们可以推断,TR, T-Jump SAXS 实验中观察到的首个转变是从一个闭合状态开始的。
此外,之前的研究已经识别出 SOSIP Env 结构的两种不同状态,包括一种开放-遮挡状态,在该状态下,每个 gp120 部分都向主三聚体轴线的外侧旋转,但是没有在 V1/V2 或 V3 区域发生重大的结构重排(如图 1B 中部所示);另一种是完全的开放状态,在这种状态下,gp120 部分不仅向外旋转,而且 V1/V2 和 V3 区域也发生了结构上的重排(如图 1B 右侧所示)。这些分析帮助我们更好地理解了 HIV-1 Env 超域在结构上的复杂动态变化。
我们首先探讨了我们的测量方法是否能够准确区分基于预融合闭合状态模型以及开放-遮挡状态或开放状态模型计算得到的理论 SAXS 差异曲线。通过对这些理论 SAXS 曲线进行奇异值分解(SVD)分析(参见图 S10C 和 S11C),我们发现了两个主要信号,这些信号与我们从 TR, T-Jump 实验中得到的差异曲线中提取的组分相一致(参见图 S8A 和 S9A)。
特别是,从闭合状态到开放-遮挡状态转变的 SAXS 差异曲线与我们通过 SVD 和 REGALS 分析得到的第二组分非常相似(见图 3C),在低散射角度处呈现出显著的负峰和正峰(见图 4A 和附加图 S12B)。而 P(r) 函数的差异曲线与 REGALS 分析中得到的组分的 P(r) 差异曲线相类似,只不过在距离轴上呈现出倒置状态(见图 4A 和附加图 S12B)。
然而,从闭合状态到完全开放状态的 SAXS 差异曲线则主要由一个单独的正峰占据,这与我们观察到的 SAXS 特征不符(见图 4B)。而开放-遮挡状态与完全开放状态之间的 SAXS 和 P(r) 函数的差异曲线与任何通过 SVD 或 REGALS 分析得到的曲线都不相似(见图 4C),这暗示了在这两种状态之间的转变要么没有发生,要么在我们的数据集中没有被明确区分出来。
这些建模分析结果表明,第二种组分的转变涉及到了一个移动到类似于已知的开放-遮挡状态的过程,在这个状态中,gp120 域从三聚体的中心轴向外旋转,但在 V1/V2 和 V3 区域并没有发生显著的结构重排。这一发现帮助我们更深入地理解了 HIV-1 Env 超域在结构上的复杂转变过程。
然后,我们深入探讨了与快速形成的中间态相关的结构变化。通过 REGALS 分析的第一个组分的 P(r) 函数差异显示,在 0 到 100 埃的范围内,散射中心显著减少,但并没有在更远的散射中心处观察到相应的增加。这表明 HIV-1 Env 蛋白在这个过程中丧失了一部分散射密度。鉴于 HIV-1 Env 是一个高度糖基化且结构动态性强的蛋白,我们考虑是否糖盾的变化可以解释通过 SVD 和 REGALS 分析得到的两个主要组分的特征。
基于无糖基化以及带有甘露糖-9(Man9)糖基化的 Env 蛋白的闭合状态、开放-遮挡状态和完全开放状态模型的理论 SAXS 分析显示,尽管两种情况下的总体特征相似,但是由于两种模型之间的粒子大小不同,导致峰值在散射向量位置上的偏移(参见图 S10D)。无糖基化模型的 SAXS 特征显示了我们感兴趣的信号,这意味着糖基本身并不是导致我们观察到的差异信号的原因。
快速形成的中间体涉及到三聚体顶点的有序到无序转变
我们继续探讨了 CH505 和 CH848 SOSIP 三聚体中通过 TR, T-Jump SAXS 观察到的中间状态是否包含了 gp120 的 V1/V2 和 V3 部分的结构重排。在 Env 蛋白的闭合状态中,V1/V2 部分与 V3 循环及 gp120 的外部区域紧密结合,并与旁边的原型体相接触(如图 1B 左侧和中间所示)。
当 Env 蛋白处于开放或半开放状态时,gp120 分子会从三聚体的中心轴线向外旋转(如图 1B 中间和右侧所示)。特别地,在完全开放状态下,V3 循环从与 V1/V2 和 gp120 外部区域紧密相连的有序状态转变为无序状态(如图 1B 右侧所示)。同时,V1/V2 区域也会从外部区域解离(如图 1B 右侧所示)。V1/V2 的这些移动导致它们从闭合状态的三β-片层结构转变为开放状态的四β-片层结构,位于 β20-β21 区域,残基编号为 418 到 445,这个结构被称作桥接片(如图 1B 右侧所示)。
这些结构上的重排导致了显著的差异,并且可能有结构中间态的存在。已知,溶液中单独存在的 gp120 部分会暴露出桥接片表位,这一区域是许多针对 CCR5 结合位的单克隆抗体(如 17b)以及针对 V3 循环的抗体(如 19b、F39F 和 3074)的靶点。
与那些不与特定单克隆抗体(mAbs)结合的稳定化 SOSIP 三聚体形成鲜明对比的是,我们通过分子动力学(MD)模拟来探索闭合状态下的 gp120 V1/V2 和 V3 区域的结构灵活性。我们从闭合状态的 CH505 SOSIP 三聚体中提取了一个 gp120 单元,以便进行模拟。这个较小的模拟系统使我们能够(i)由于系统中的原子数量减少,模拟更长的时间尺度;以及(ii)由于缺少原型体间的相互作用,更清楚地观察到 V1/V2 和 V3 区域的 gp120 动态。
通过在 50°C 的系统温度下运行的 250 条独立的 5-微秒 MD 模拟轨迹,我们研究了 V1/V2 和 V3 构象的变化。相对于闭合状态,开放状态的 V1/V2 和 V3 构象的根均方偏差(RMSDs)分别为 6.5 和 8.2 埃。相比于初始闭合结构,V1/V2 和 V3 的平均 RMSDs 分别为 2.1 ± 0.8 埃和 1.3 ± 0.4 埃,而构象稳定的 gp120 外域 β-片层的平均 RMSD 为 0.9 ± 0.2 埃。V1/V2、V3 和 β20-β21 的最大 RMSD 观测值分别为 6.1、4.6 和 2.1 埃。在模拟中,我们并未观察到 V1/V2 的明显位移或者在 β20-β21 中的链条转换。这些发现为我们提供了闭合状态下 gp120 结构变化的深入视角。
虽然 V1/V2 和 V3 部分并没有展现出明显向开放或类似开放状态转变的迹象,我们却发现在闭合状态的三聚体中,负责形成原型体顶点接触的环发生了从有序到无序的转变。这引发了我们的一个疑问:顶点的这种有序到无序的转变是否能解释快速形成的中间态中所观察到的散射变化。
根据 REGALS 分析的第一个组分 P(r) 函数所示的散射密度的减少,这种变化与有序到无序的转变是一致的。这种变化是由于密集蛋白质折叠与水合良好的无序链之间的密度差异减小所致。
因此,我们进一步探讨了一个缺少 gp120 之间接触环的结构是否能产生我们所观察到的散射差异曲线和 ΔP(r) 函数。针对缺少特定残基(160至171和306至317)的模型所计算出的散射和 P(r) 函数的差异轮廓与观察到的 REGALS 第一组分紧密匹配(见图 4E 和附加图 S12A)。
这表明散射密度的减少,很可能指向这部分三聚体转变为无序状态。总的来说,这些发现支持了一个模型,即 T-Jump 引起的转变导致三聚体顶点处的接触消失,以及 gp120 分子向一个开放-遮挡状态的旋转。
原型体间的二硫键稳定了闭合的 Env 三聚体
TR, T-Jump SAXS 实验的发现揭示了一个重要现象:即便在那些不显著结合开放状态特异性抗体的 Env SOSIP 中,向更开放的构形转变的过程依然可能发生。一项近期的猕猴免疫学研究显示,尽管在预融合闭合状态下对免疫原进行了稳定处理,但仍然能够诱导产生针对开放-遮挡状态的抗体,这与我们的发现相吻合。我们对 SAXS 数据进行的建模分析强调了 gp120 之间在顶端接触点的变化对于控制 gp120 旋转的重要性。因此,为了阻止 Env 蛋白向开放-遮挡构形的过渡,很可能需要阻断顶端区域的结构重排。
基于这个目标,我们综合运用了多种计算工具来寻找可能稳定原型体顶端的突变。通过这一分析,我们在 CH505 SOSIP 三聚体的设计基础上找到了一种 V127C-D167C 的二硫键突变,该突变能够有效地在 gp120 原型体之间的 V1/V2 接触区域形成“钉扎”,增强结构稳定性。此外,我们还应用了先前报道的 F14 和 SOSIP 2P 突变来进一步稳定这种设计,旨在提升其表达和正确折叠的能力,并通过引入一个 N197D 糖基删除突变来增强 CD4结合位抗体的结合效果。通过负染电子显微技术,我们确认了 Env 三聚体的闭合构形,为 Env 蛋白的结构研究和疫苗设计提供了新的见解。
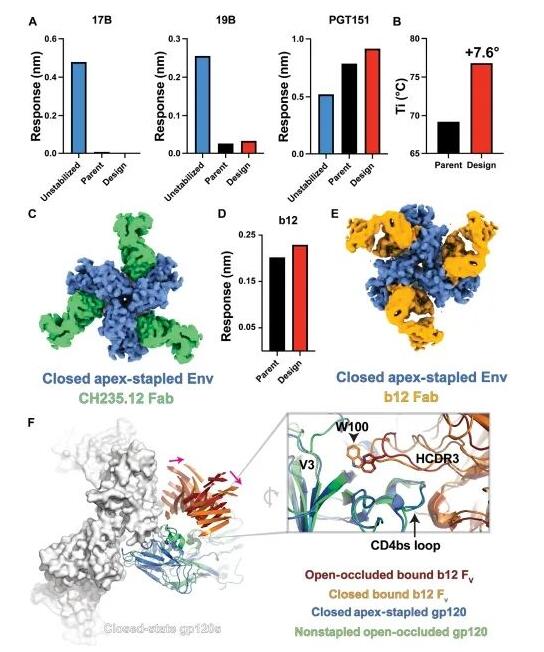
SDS-聚丙烯酰胺凝胶电泳(SDS-PAGE)的分析和与 19b 抗体的结合测试显示,我们准备的 Env SOSIP 样品中存在一部分因原型体间二硫键形成不当而暴露了 V3 表位的群体。通过对绑定了 CH235.12 抗体的 CH505 顶点钉扎 SOSIP 设计进行的单颗粒冷冻电子显微(cryo-EM)分析,我们发现了两种不同状态的存在,一种是闭合状态,另一种是 V1/V2 顶端区域缺失清晰定义的密度状态。
为了排除这种开放状态,我们采用了针对 V3 循环的 3074 抗体进行了负向筛选,以进一步纯化 Env SOSIP 样本。这一步骤成功去除了 SDS-PAGE 中未通过原型体间链接形成的条带,减少了对 19b 抗体的结合,并提高了对 trimer 特异性的 PGT151 抗体结合的亲和力。通过这种顶点钉扎设计,我们观察到热变性温度的显著提高(提高了 7.6°C),显示出更好的稳定性。
这种通过原型体间二硫键钉扎的闭合状态三聚体,在原型体顶端环接触区域的密度分析中显示了较低的分辨率,这可能反映了该区域构象的多样性。尽管如此,在较低的映射轮廓水平上,我们依然能够观察到与原型体间形成二硫键一致的密度。这些研究结果共同表明,通过在三聚体顶点的原型体间形成二硫键,我们能够有效地增强 Env 三聚体的稳定性。
图5 原型体间的二硫键帮助稳定 Env 三聚体的闭合状态。(A)这部分研究检测了不同版本的 CH505 SOSIP 对一系列抗体的结合反应,包括原始的 CH505 SOSIP、经过稳定突变改良的 CH505 SOSIP,以及通过原型体间二硫键钉扎改良的 CH505 SOSIP。这些抗体分别是针对共受体结合位的 17b、针对 V3 循环的 19b,以及针对闭合状态三聚体顶点的 PGT145。(B)对比了原始 CH505 SOSIP 和通过原型体间二硫键钉扎的 CH505 SOSIP 在热变性测试中的温度拐点,显示了后者在热稳定性上的提高。(C)展示了与 CH235.12 Fab 结合的二硫键钉扎 CH505 SOSIP 的高斯滤波图,用于进一步分析这种结构的细节。(D)检测了经过稳定处理的 CH505 SOSIP 和通过原型体间二硫键钉扎的 CH505 SOSIP 与 b12 Fab 的结合反应,观察它们之间的互动。(E)提供了与 b12 Fab 结合的二硫键钉扎 CH505 SOSIP 的高斯滤波图,为深入理解这一交互提供了视图。(F)左图比较了闭合状态下与 b12 抗体结合的 CH505 SOSIP 设计与 B41 分离物的开放-遮挡状态,特别强调了 b12 Fv 部位的位置变化。右图则对比了 b12 结合的顶点钉扎 CH505 SOSIP 设计与 B41 分离物 gp120 域之间 HCDR3 位置的不同。
这些发现表明,在 Env 三聚体顶点形成的原型体间二硫键可以有效防止蛋白质向开放-遮挡状态或中间状态的转变,从而稳定其闭合构形,这对于理解 HIV Env 的结构动态及其在疫苗设计中的应用具有重要意义。
我们在这里揭示了 CH505 和 CH848 DS Env SOSIP 结构能够在微秒级时间内转变成类似开放-遮挡状态。CH505 的研究结果指出,转变到类似开放-遮挡状态之前,存在一个快速形成的中间阶段。
然而,由于模型拟合 CH848 数据时的不确定性,我们不能断定 CH848 DS SOSIP 中也存在这样一个快速形成的中间体。即便如此,模型的比较还是支持了一个关于 CH848 转变的假设,即其经历的中间态转变和开放-遮挡转变都比 CH505 来得更快。
通过 gp120 的分子动力学(MD)模拟,我们发现,除了原型体间接触的 gp120 环之外,其起始的闭合状态结构在快速形成的中间体出现的时间尺度上是稳定的。在模拟中,这些环展现了从有序到无序的转变。基于这一点,我们探索了一个涉及顶端环接触点处散射密度丧失的中间体形成的模型。这个模型与我们通过 SVD 和 REGALS 分析得到的结果一致,表明这些环经历了从有序到无序的转变。
第二个较慢的转变与 CH505 和 BG505 SOSIP Env 样品之间的散射轮廓差异及开放状态抗体的结合相一致。因此,这个状态很可能是指 gp120 向外旋转,从而增加了 gp120 顶端残基间距的 Env 结构。然而,基于现有的散射数据,我们还无法确定 V1/V2 和 V3 的重排程度,或者 gp120 旋转的具体程度。
值得注意的是,虽然我们使用的 Env SOSIP 经过了彻底的纯化和特性分析,但我们仍不能完全排除样品中可能存在多种未被识别的折叠形式的可能性。这可能意味着观察到的两种不同的转变速率并非顺序发生的事件,而是与两种不同折叠状态之间的相对转变倾向相关。
对于我们设计的顶点钉扎版本,通过冷冻电子显微技术的三维分类,我们容易识别出不同的折叠状态群体。在我们研究的其他相关 SOSIP 结构中,并没有发现这样的状态,也没有发现任何表明折叠不稳定的批次间质量控制的显著变化。
如果存在不同的折叠形式,理论上会导致不同的热变性曲线,从而期望看到明显的双峰或多峰变性曲线,但在我们的样品中并未观察到这种情况。因此,我们认为,我们观察到的转变确实与单一折叠形式的连续事件相关。
最后,要准确量化我们观察到的末态中 gp120 旋转的范围及其构象动态是有挑战的。为了深入理解这些结构变化,需要进一步实验,比较在已知构象状态下锁定的三聚体的 SAXS 测量结果与其他结构数据。
本研究揭示了 Env 三聚体从闭合状态过渡到 gp120 结构域向外旋转、远离三聚体中心的状态,是一个按步骤进行的过程。不是 gp120 的旋转与三聚体顶端接触点的断裂同时发生,而是 gp120 顶端的环首先变得无序化。
我们的研究结果显示,这种无序化发生而并不需要 V1/V2 完全重排,这通常会导致桥接片的形成。这一发现与之前的研究相符,后者表明,V3 循环的暴露可以在不暴露桥接片的情况下发生。与 CH505 Env SOSIP 的有序到无序转变相比,向更开放的构型过渡的时间尺度几乎长出两个数量级。一旦三聚体顶端的接触点被打破,这种过渡的控制就必须依赖于 gp120 和 gp41 之间剩余的接触点。
在达到平衡状态时,这些过渡的顺序排列形成了一个动力学开关,控制着 gp120 旋转的发生,这取决于顶端由有序到无序的转变和 gp120 与 gp41 之间剩余接触点断裂的联合概率。这些过渡概率的变化很可能解释了 CH848 的时间常数降低,并可能解释不同 Env 变体之间开放状态出现的概率差异。最终,这些在微秒级时间尺度上的过渡速率差异,可能决定了 Env 对受体诱发的构型变化的敏感度。
我们在这里发现的 Env 结构转变与 HIV-1 的全长 gp160 Env 之间的具体联系还是一个待解的问题。天然的 Env 三聚体是嵌入在细胞膜中的,这可能会影响 Env 不同结构状态之间的构象转变及其动力学特性,尤其是与我们在此研究的可溶性 gp140 构造体中测量的转变相比较。
已知膜嵌入式的 gp160 Env 能够在多种结构状态之间进行转变,这与可溶性的 SOSIP gp140 构造体相似(2, 17, 18, 73),并且 gp160 与 gp140 SOSIP 结构之间也存在结构上的相似性(74, 75)。
因此,我们这里呈现的研究结果很可能对理解 gp160 Env 也具有重要意义。然而,具体如何将我们观察到的状态与以前发现的状态联系起来还不清楚,这主要是由于缺少跨膜域和在这些构造体中添加的三聚体稳定化 SOSIP 突变。这是在疫苗接种背景下,对于理解抗体如何可能利用 Env 结构超出预融合闭合状态的易变性的关键问题。
这些状态在 CD4 受体结合前在二级病毒中极为罕见。这对疫苗接种的影响有两方面:更可能激发出针对开放-遮挡状态的低效中和抗体,同时,具有广泛中和潜力的抗体可能倾向于获得支持这些状态的突变,而不是那些对提升中和功能至关重要的突变,这限制了它们发展成具有广泛中和能力的抗体。
我们在这里展示的顶点钉扎设计通过阻止 gp120 的活动,从而排除了这些不良影响。与抗体相互作用效果一致,b12 抗体的 NSEM 和冷冻电镜结构揭示了,与未进行钉扎处理的构建相比,顶点钉扎显著改变了抗体的接近角度。
总的来说,这些发现证明了仅在微秒级时间尺度上可观察到的瞬时中间状态对于控制 HIV-1 Env 结构起着关键作用。在疫苗开发中,阻断这些早期的状态转变是关键的,这有助于确保正在成熟的抗体能够继续发展其中和广度的潜力。
如何生产重组HIV-1 Env SOSIP
我们使用了一种特殊的嵌合体,它结合了 CH505 gp120 和截短版的 BG505 gp41 结构域,包括一种经过特殊处理以增强稳定性的 CH505TF SOSIP,以及从 HIV 感染者 CH505 在感染后第 53、78 和 100 周收集的样本中分离出的 CH505 SOSIP。
此外,我们还研究了一种经过 DS 处理以提高稳定性的 CH848 第 949 天的 SOSIP,以及一个 N332 BG505 SOSIP。这些都在 Freestyle 293-F 细胞中进行表达,这是一种专门用于生产此类蛋白的细胞系。
在开始转染过程之前,我们首先将细胞在特定的表达培养基中稀释到适当的浓度,并将其准备在 950 毫升的体积中。接下来,我们将质粒 DNA(包含 SOSIP 和 furin 的表达序列)以一定比例混合,并使用特定的转染试剂和培养基进行处理,以促进复合体的形成。这个混合物随后被添加到细胞培养中,在特定的温度和 CO2 浓度条件下孵育 6 天。
在孵育期结束时,我们通过离心来收集细胞上清,然后通过特定孔径的过滤器进行过滤,最后使用一种特殊的横流过滤设备将其浓缩,以便于后续的实验分析。这一系列精心设计的步骤确保了我们能够高效且准确地生产出所需的重组 Env SOSIP 蛋白。
信封 SOSIP 的纯化过程是通过一个特别设计的 PGT145 亲和色谱柱进行的,该柱子已经用 15 mM 的 Hepes 和 150 mM 的 NaCl(pH 值为 7.1)溶液进行了平衡。我们通过将 PGT145 单克隆抗体与一种特殊的 Sepharose 4B 树脂(由 GE Biosciences 提供)结合,并将其装填入一个 Tricorn 色谱柱(由 GE Healthcare 提供)来创建这个亲和柱。
然后,我们将细胞上清以每分钟 2 毫升的速度通过这个柱子,使用 AKTA go 色谱系统(由 Cytiva 生产)来进行操作,并通过使用 15 mM 的 Hepes 和 150 mM 的 NaCl 进行三次柱体积的清洗。
接着,我们使用 3 M 的 MgCl2 从色谱柱中洗脱蛋白,并将其在含有 15 mM Hepes 和 150 mM NaCl 的缓冲液中稀释。之后,我们通过使用一种具有 100-kDa 分子量截止值的 Amicon Ultra-15 离心过滤单元(由 Millipore Aldrich 提供)进行超滤,将蛋白样本转换到新的缓冲液中,并将其浓缩至少于 0.5 毫升,以备进行凝胶过滤色谱分析。
最后,我们在 AKTA go 系统上使用一个 Superose 6 10/300 GL 色谱柱(由 Cytiva 提供)在 15 mM 的 Hepes 和 150 mM 的 NaCl 中进行凝胶过滤色谱分析。我们收集了包含三聚体 SOSIP 的那些部分,这一系列精细的步骤确保了我们能够高效地纯化出所需的蛋白。
我们对所有生产的 Env SOSIP 批次进行了一系列质量控制测试,确保我们得到的 Env 三聚体结构正确且稳定。这些测试包括分析性的凝胶过滤色谱、SDS-PAGE 蛋白质电泳、热变性分析、生物层析技术(BLI)和负染电子显微镜(NSEM)。我们设定了一个标准,即任何在这些测试中的结果偏离平均值超过一个标准差(SD)的批次都不会被用于后续实验。
特别是对于 TR, T-Jump SAXS 实验,我们将所有的 CH505 SOSIP 批次合并起来进行实验。实验完成后,我们对这个合并后的样本再次进行上述质量控制测试,以确保实验过程中样本的质量和完整性没有受到影响。
重组抗体和抗体 Fab 生产
我们在特殊的 Expi293 表达系统中利用 Expi293F 细胞生产了抗体(来自 Thermo Fisher Scientific,产品编号 A1435101)。首先,我们将细胞浓度调整到每毫升 1.25亿个,总体积为 100 毫升。
接着,我们将重链和轻链的质粒 DNA 以 1比1的比例混合(每次转染 100 毫升用 50 微克),并在 Opti-MEM I 低血清培养基(来自 Thermo Fisher Scientific,产品编号 31985062)中与 Expifectamine 293 转染试剂(来自 Thermo Fisher Scientific,产品编号 A14525)共同孵育,以形成 DNA-试剂复合体。
然后,我们将这种混合物加入已经准备好的细胞培养液中,并将其放置在一个摇床上,在 37°C 和 8% 的 CO2 环境中孵育 6 天,摇动速度为每分钟 120 转。到了第 6 天,我们通过以 4500 转/分钟的速度离心 45 分钟来收集细胞的上清液。最后,我们用一个 0.45 微米的 PES 过滤器对上清液进行过滤。
我们采用了 Cytiva HiTrap Protein A HP 抗体纯化柱(Cytiva,产品编号 29048576)对每种抗体进行纯化,该纯化柱预先用 1×磷酸盐缓冲溶液(PBS)进行平衡处理。我们将收集的上清液以 0.5 毫升/分钟的速度通过已经平衡好的纯化柱,此过程使用的是 Cytiva 公司的 AKTA start 色谱系统,之后进行了三次使用 1×PBS 的洗涤步骤。
接着,抗体通过 Pierce 公司的免疫球蛋白 G(IgG)洗脱缓冲液(pH 值为 2.8,Thermo Fisher Scientific,产品编号 21004)进行洗脱。样品的缓冲液通过添加 tris(pH 值为 9)以 100 微升/毫升的比例进行中和处理(Thermo Fisher Scientific,产品编号 J60707)。生产出的所有批次都经过了包括 SDS-PAGE 和热变性分析在内的质量控制,以确保其品质。
热变性分析
进行热变性测试是为了评估 Env SOSIP 外域的稳定性,这项测试通过 Tycho NT.6 设备(来自 NanoTemper Technologies)完成。我们首先将 Env SOSIP 样本稀释到 0.15 毫克每毫升的浓度,在 pH 值为 7.1 的含有 150 mM NaCl 的 15 mM Hepes 缓冲液中。
接着,我们在样本从 35°C 加热至 95°C 的过程中,以每分钟 3°C 的速度,记录了在 330 和 350 纳米波长下的内在荧光值。通过 Tycho NT.6 设备,我们计算了荧光比值(350/330 纳米)和样品的拐点温度(Ti),这些数据帮助我们了解样品在加热过程中的稳定性变化。
生物层析技术
我们通过生物层析技术(BLI,使用 OctetR4 设备,来自 FortéBio)来测试样本中的单克隆抗体(mAb)和 Fab 片段与目标的结合能力。首先,我们把抗体固定到专门的传感器尖端上,这是通过将其在含有 4.5 微克/毫升抗体的 pH 7.1、15 mM Hepes 缓冲液和 150 mM NaCl 溶液中浸泡 300 秒实现的,之后在同样的溶液中以 1000 rpm 速度洗涤 60 秒。
对于 Fab 片段,我们采用了相同的方法但使用了蛋白 A 传感器尖端。之后,这些准备好的传感器尖端被放入含有 SOSIP(45 微克/毫升)的样品中,持续 180 秒。我们所报告的结合数据是在结合反应阶段结束时测得的。所有数据都通过 FortéBio 提供的 Octet 数据分析软件进行了处理和评估。
负染电子显微镜
利用负染电子显微技术对样品进行观察,首先将样品在室温下稀释到 20 微克/毫升,使用了一种特殊的混合溶液(含有 0.1% 的 n-十二烷基 β-d-麦芽糖苷、50 mM 的 Tris 缓冲液(pH 7.4)、150 mM 的 NaCl 和 0.03 mg/ml 的去氧胆酸钠)。
接着,我们迅速将样品滴加到一块经过特殊处理以增加附着力的碳涂层电镜网格上,并保持 8 至 10 秒。之后,我们使用尿素甲酸盐溶液(浓度为 2 克/分升)对样品进行染色1分钟,随后吸去多余的液体,并让网格在空气中自然干燥。
干燥后的网格在一个 Philips EM420 电子显微镜下进行了检查,该显微镜的工作电压为 120 kV,放大倍数约为 49,000 倍。我们在每像素 2.4 埃的分辨率下,通过一个 7600 万像素的电荷耦合设备(CCD)摄像头收集了 114 幅图像。利用 Relion 3.0 软件按照标准流程对这些图像进行了二维分类平均分析,以便更清晰地观察样品的结构特征。
TR, T-Jump 小角 X 射线散射光束线设置
在阿贡国家实验室的先进光源设施中,我们使用 BioCARS 14-ID-B 光束线来开展了温度跳变小角度 X 射线散射(TR, T-Jump SAXS)实验和一系列固定温度下的 SAXS 实验。为此,我们将 CH505 蛋白在 pH 值为 7.1 的 Hepes 缓冲液中进行了浓缩,该缓冲液含有 150 mM 的 NaCl,最终得到大约 4 毫升的蛋白溶液,浓度为 5.125 毫克/毫升。同时,我们还准备了两种不同浓度的 CH848 蛋白溶液,同样使用 pH 为 7.1 的 Hepes 缓冲液进行浓缩,得到大约 3 毫升的 1.66 毫克/毫升和大约 2 毫升的 1.4 毫克/毫升溶液。
在实验使用前,所有蛋白样品都经过了 0.22 微米过滤器的过滤和脱气处理。我们用一个蠕动泵将蛋白和缓冲液混合物通过一个定制的、温度可控的氮化铝支架上的 700 微米石英毛细管注入。通过使用 7 纳秒的红外激光脉冲(波长为 1.443 微米,每脉冲能量为 1.1 毫焦)来触发样品的温度跳变,此过程旨在激发水分子中的 O-H 键伸缩。
在一定的时间延迟后,我们用 1.6 微秒(11束模式)或 3.6 微秒(24束模式)的粉红色 X 射线束来探测样品,这些 X 射线束的光子能量为 12 keV,带宽为 300 eV。最后,我们使用 Rayonix MX340-HS X 射线探测器收集了一系列在 0 至 2.5 埃的散射向量(q)范围内的图像。
T-Jump 校准
T-Jump(温度跳变)的强度是通过一种之前已经描述过的方法来校准的。具体来说,我们测量了在 25°到 51°C 范围内对于 CH505 缓冲液以及在 30°到 45°C 范围内对于 CH848 缓冲液的静态小角度 X 射线散射(SAXS)信号。对于这两组数据,我们把温度跳变的结果和静态测量的结果结合起来,然后对这些数据进行了奇异值分解(SVD)处理。在这个过程中,我们发现第二主要成分的左向量展现出了与缓冲液信号差异相匹配的特征。
基于这一发现,我们对静态温度下的数据进行了线性回归分析,特别关注第二主要成分的右向量。通过这种分析,我们能够根据回归线的斜率和截距来确定 T-Jump 实验中温度变化的具体幅度。这样的校准方法帮助我们精确控制和理解实验中的温度变化,从而确保实验结果的准确性和可重复性。
静态 SAXS
静态小角度 X 射线散射(SAXS)数据收集工作是在劳伦斯伯克利国家实验室的先进光源设施中的 SIBYLS 12.3.1 光束线完成的,这项工作关注了 BG505 和 CH505 Env SOSIP 样品。为了确保样品的质量,我们在 -79°C 的条件下通过隔夜快递将样品运至实验室,并在测量前将其保存在 -80°C。
我们对一系列不同浓度的样品进行了测试,浓度范围从 0.5 毫克/毫升到 3 至 5 毫克/毫升,每个样品的总曝光时间为 10 秒。随后,我们使用 ATSAS 软件套件对收集到的数据进行了处理和分析,以获得详细的散射轮廓,这对于理解 Env SOSIP 样品的结构特性至关重要。
静态SAXS温度序列
静态SAXS温度序列数据是在先进光子源BioCARS 14-ID-B光束线上测量的,该光束线用于上文描述的TR, T-Jump SAXS。收集了15 mM Hepes缓冲液和CH505 Env样品(5.125 mg/ml)在25°, 35°, 44°和50°C(见图S3)的静态SAXS轮廓。对于CH848 Env样品,在30°, 35°, 40°和44°C(见图S4)收集了15 mM Hepes缓冲液、CH848(1.66 mg/ml)和CH848(1.4 mg/ml)的静态SAXS轮廓。静态SAXS信号是在BioCARS 14-ID-B光束线上,使用24束连续平移模式以20 Hz和每脉冲3.6 μs,197脉冲每图像曝光时间收集的。每组数据共收集了50张图像,散射向量范围q = 0到2.5 Å−1。
CH505和CH848时间延迟系列
在先进光子源BioCARS 14-ID-B光束线上收集了TR, T-Jump SAXS数据。对CH505 Env SOSIP样品(5.125 mg/ml)在由IR脉冲T-Jump从44°加热到约50°C后的几个不同时间延迟进行了数据收集:500 ns, 1.5 μs, 3.0 μs, 5.0 μs, 10 μs, 50 μs, 100 μs, 500 μs, 1 ms, 10 ms和100 ms。对CH848 Env SOSIP样品(1.66和1.4 mg/ml)收集了时间分辨SAXS数据,时间延迟为5 μs,10 μs, 50 μs, 100 μs, 250 μs, 500 μs, 750 μs, 1 ms, 10 ms和100 ms。
对于每个测量的时间延迟,也在IR激光加热前的-10和-5μs测量了TR, T-Jump SAXS(“激光关闭”;见图S5A)。为了适应时间尺度的大变化并最小化由于实验漂移导致的系统误差,我们对缓冲液和Env进行了多组测量,每组之间有重叠的时间延迟。CH505 Env SOSIP TR, T-Jump SAXS在三组中测量。
CH505 Env第1组包括-10 μs, -5 μs, 10 μs, 50 μs, 100 μs, 500 μs和1 ms,在20 Hz的连续平移模式下使用24束进行测量,每个脉冲3.6 μs和197脉冲每张图像(65),为每个时间延迟收集250张图像。CH505 Env SOSIP的第2组在20 Hz连续平移模式下使用11束测量,每个脉冲1.6 μs和197脉冲每张图像(65),包括-10, -5, 1.5, 3和5μs,每个时间延迟收集362张图像。为CH505 Env第3组收集了200张图像,该组在5 Hz阶跃平移模式下使用24束测量,每个脉冲3.6μs和197脉冲每张图像(65),包括时间延迟-10 μs, -5 μs, 1 ms, 10 ms和100 ms。
使用单束在20 Hz连续平移模式下(65)分别收集了500-ns时间延迟的25张图像,T-Jump从42°C开始。CH848数据在三组中收集。CH848 Env第1组包括-10 μs, -5 μs, 5 μs, 10 μs, 50 μs, 100 μs, 500 μs和1 ms,在20 Hz的连续平移模式下使用24束进行测量,每个脉冲3.6 μs和197脉冲每张图像(65),为每个时间延迟收集100张图像。
CH848 Env SOSIP的第2组在20 Hz连续平移模式下使用24束测量,每个脉冲3.6 μs和197脉冲每张图像(65),包括-10 μs, -5 μs, 5 μs, 10 μs, 50 μs, 100 μs, 250 μs, 500 μs, 750 μs和1 ms,每个时间延迟收集100张图像。为CH848 Env SOSIP第3组收集了100张图像,该组在5 Hz阶跃平移模式下使用24束测量,每个脉冲3.6 μs和197脉冲每张图像(65),包括时间延迟-10 μs, -5 μs, 1 ms, 10 ms和100 ms。
建模
使用Modeller10.1(68)构建了CH505和CH848 Env SOSIP在闭合(Closed)、闭合但无顶端接触(Closed_NoApex)、开放-遮挡(OpenOccluded)和开放(Open)构象的原子模型。模型使用的模板包括蛋白质数据银行(PDB)ID 6UM6(71)用于闭合的原型体,PDB ID 6CM3(13)用于开放的原型体,以及PDB ID 7TFN(15)用于开放-遮挡的原型体。使用Clustal Omega在DNASTAR MegAlign软件中进行CH505或CH848 Env SOSIP与模板之间的序列比对。在Modeller脚本中添加了二硫键补丁,以确保形成二硫键。
使用Glycosite(69)来预测CH505和CH848 Env SOSIP中可能的N-连结糖基化位点,并使用Glycosylator(80)对每个模型进行Man9糖基化。进行了三轮细化以消除糖基冲突。所有细化轮次均使用了10次迭代、0.01的变异率和30的种群大小。前两轮细化使用了10代,而最后一轮细化使用了20代。每次细化之间个体数(80)减少了两个。
使用Glycosite(编号69)工具预测了CH505和CH848 Env SOSIP中可能存在的N-连结糖基化位点,并且使用Glycosylator(编号80)工具对每个模型进行了Man9糖基的添加。为了消除糖基冲突,进行了三轮细化处理。所有细化过程均采用了10次迭代、0.01的变异率和30的种群大小。前两轮细化采用了10代,而最后一轮细化采用了20代。每经过一轮细化,个体数(80)减少两个。
针对每个糖基化模型的理论SAXS轮廓,是通过FoXS服务器(编号81)计算得出的。这些理论SAXS散射轮廓的P(r)分布在ATSAS Primus软件(编号67)中确定,通过从闭合P(r)曲线中减去开放或开放-遮挡P(r)曲线,计算出ΔP(r)曲线,并与实验及REGALS SAXS轮廓和ΔP(r)曲线进行比较。
对闭合、开放-遮挡和开放模型的理论散射曲线进行了SVD分析,包括带有Man9糖基和未糖基化的情况,以提取这些结构之间转换的组分信号。在ATSAS Primus软件(编号67)中对每个理论SAXS轮廓进行了Guinier分析和配对距离分布,以确定Rg(表S5)。
分子动力学
使用CHARMM-GUI糖基模型器(82–87)根据glycosite预测的(70)糖基化位点,用Man5糖基对CH505模型基于PDB ID 6UDA的gp120系统进行糖基化处理,为模拟做准备。对闭合的gp120在50°C下运行了总共250个,5微秒的轨迹。使用Amber22软件包配合pmemd CUDA实现(88–90),在CHARMM36力场(91–94)和TIP3P水模型(95)下进行模拟。
使用150-Å的八面体盒子,以便在模拟过程中允许V1/V2和V3位移。两个系统都用150 mM NaCl中和。首先进行了一万步能量最小化,包括1000步最陡下降法最小化,对蛋白施加500 kcal/mol·Å的约束。其次是不受约束的能量最小化,包括一万步和1000步最陡下降步骤。最小化后,进行了20 ps在恒定体积下加热,对蛋白原子施加10 kcal/mol·Å的约束,然后是5 ns无约束的平衡,保持恒定温度323.15 K,使用Langevin温控器(96),并以各向同性位置缩放保持1 atm压力。
进行了250纳秒无约束的NVT集合模拟以平衡系统,随后在NVT集合中进行了5微秒的生产模拟。电荷相互作用通过粒子网格Ewald方法(97)计算,截断半径为12 Å,转换距离为10 Å。使用SHAKE算法(98)和氢原子质量重分配(99)约束氢原子,并允许4-fs时间步长。
使用AmberTools21中的cpptraj工具(100)确定V1/V2(HXB2分离株编号中的残基129至133、135至150、154至163、168至177、181至183和190至193)、V3(HXB2分离株编号中的残基297至308和311至319)和gp120核心(HXB2分离株编号中的残基288至291、280至284、355至358、370至374、377至382、411至416、439至452和462至466)β片层α碳的RMSD时间序列。使用自定义Python脚本确定所有复制品聚集在一起的平均RMSD和频率分布。
基于理性结构的设计
CH235.UCA结合的CH505.M5.G458Y SOSIP三聚体结构(6UDA)在Maestro(101)中使用蛋白质准备向导(102)准备,随后通过使用Schrödinger的半胱氨酸突变(103)和残基扫描(104)工具进行了计算机辅助突变分析。
首先对V1/V2和V3区域的互原型体接触残基进行了残基扫描。通过得分和视觉检查用于选择准备的构建物。所选设计是在先前报道的CH505分离株SOSIP骨架(76)中准备的,引入了表达增强的2P突变(60)、禁用变构机制的一组突变F14(48)和增强CD4结合位抗体结合的N197D突变(60)。
冷冻电镜样品准备、数据收集和处理
纯化的信封SOSIP外域制备品在pH 7.1的15 mM Hepes缓冲液中,含150 mM NaCl,浓度调至4至5 mg/ml,并与Fab以1:5的摩尔比混合。将2.5 μl的复合物滴定在经过15秒辉光放电处理的CF-1.2/1.3网格上,使用PELCO easiGlow辉光放电清洁系统。
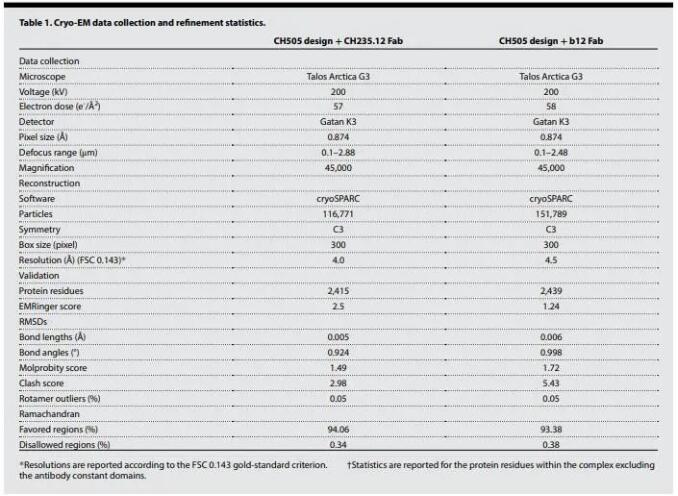
在大于95%的湿度中孵育30秒后,吸去多余的蛋白质,使用Leica EM GP2冲冻仪(Leica Microsystems)在液态乙烷中快速冻结。冷冻网格在装备有K3探测器(Gatan)的Talos Arctica(Thermo Fisher Scientific)中成像。单个帧被对齐、剂量加权并进行对比传递函数(CTF)校正,随后进行粒子选取、二维分类、原始模型生成、异质细化、同质三维细化以及cryoSPARC中的局部分辨率计算(表1)(105)。
冷冻电镜结构拟合和分析
使用CH505.M5.G458Y SOSIP结构(6UDA)和CH235.12(5F96)或b12(2NY7)结构,来拟合冷冻电镜图谱,在ChimeraX(106)中进行。在PyMol(107)中进行突变操作。然后使用Isolde(108)进行坐标拟合,随后通过使用Phenix(109)的实空间细化进行迭代细化。结构和图谱分析使用PyMol、Chimera(110)和ChimeraX(106)执行。
注释:
HIV:人类免疫缺陷病毒(Human Immunodeficiency Virus),是一种病毒,能够攻击人体的免疫系统。
SOSIP:是指一种特定构造的HIV-1 Env蛋白,用于研究和疫苗开发中。SOSIP三聚体能够模拟HIV病毒表面上的自然形态,有助于研究病毒如何与宿主细胞结合和进入。
CH505和CH848:这些是特定的HIV Env SOSIP变体的代号,代表不同的病毒株或从不同患者分离得到的病毒样本。
PDB:蛋白质数据银行(Protein Data Bank),是一个存储三维结构数据的数据库,用于生物分子,尤其是蛋白质、核酸和复合物。
SAXS:小角X射线散射(Small-Angle X-ray Scattering),是一种用于研究材料结构的技术,特别适用于蛋白质和其他生物大分子的形态和大小分析。
cryo-EM:冷冻电子显微镜技术(Cryogenic Electron Microscopy),一种先进的显微镜技术,可以在近原子级分辨率下观察生物大分子的结构。
Fab:抗体片段(Fragment antigen-binding),是抗体的一部分,负责与抗原特异性结合。
信息来源:SCIENCE ADVANCES 2 Feb 2024 Vol 10, Issue 5 DOI: 10.1126/sciadv.adj0396 Microsecond dynamics control the HIV-1 Envelope conformation
信息来源:【互联网】
排版编辑:【高翔】
信息发布:【阿杰】
项目支持:【和布克赛尔县】
联系方式:13999198395 QQ\微信:137655 公益转编,侵删
新疆乌鲁木齐艾滋病检测、艾滋病阻断、HIV检测、HIV阻断、公益组织、感染者咨询 电话13999198395
| 姓名 |
电话 |
| HIV药物阻断 |
|
| HIV药物预防 |
| 疫情期间HIV药品借药援助 |
| 采血结果查询 |
|
| HIV检测 |
|
| A友健康互助群 |
|
 |
 |
|